製品・サービス情報
|
|
JS Link 社 DNA メチル化アレイ受託解析
InfiniumTM MethylationEPIC BeadChip 受託解析サービス |
|
 |
illumina 社の DNA メチル化ビーズアレイ「Infinium® MethylationEPIC v2.0 BeadChip」を用いた受託解析サービスです。
DNA メチル化は、遺伝子発現の制御において重要な役割を果たしています[1, 2]。illumina 社では 10 年以上にわたり、ゲノムワイドな
DNA メチル化を定量的に測定するための BeadArrayTM technology を搭載した堅牢なマイクロアレイベースのツールを研究者に提供してきました。これまで Infinium HumanMethylation450
および Infinium MethylationEPIC v1.0 BeadChip は、エピゲノムワイド関連研究(EWAS)のデータ収集のために研究コミュニティによって使用され、がん研究[3,
4]、遺伝性疾患[5]、老化[6]、分子疫学[7]などの分野で、メチル化に基づくバイオマーカーの発見と応用を可能にしました。
新たに更新された Infinium MethylationEPIC v2.0 BeadChip は、信頼性の高い Infinium ケミストリーの基盤の上に構築され、エピジェネティクス研究の新時代において、より多くの生物学的発見を可能にするために、専門家が選択した強化されたコンテンツを提供します。
*本サービスは、JS Link 社(韓国)との業務提携により、サービス提供いたします。 |
 Infinium® MethylationEPIC v2.0 BeadChip の特長 Infinium® MethylationEPIC v2.0 BeadChip の特長
|
InfiniumTM MethylationEPIC v2.0 BeadChip Kit は、サンプルのプールやインデックス作成を必要としない、合理化されたワークフローに従っています。8
サンプルフォーマットの BeadChip は、Infinium HD メチル化アッセイに従って処理され、iScanTM でスキャンされます。
CpG アイランド、遺伝子、エピゲノムワイド関連解析用エンハンサーを広範囲にカバーする頑強なメチル化プロファイルマイクロアレイ
 |
 |
| 図1. InfiniumTM MethylationEPIC v2.0 BeadChip と iScanTM |
InfiniumTM MethylationEPIC v2.0 BeadChip は、エンハンサー領域,
遺伝子本体、プロモーター、および CpG アイランドにおける 935,000 以上の CpG を特徴としています。 |
 |
【ゲノムワイドな最先端コンテンツ】
|
InfiniumTM MethylationEPIC v2.0 BeadChip は、InfiniumTM MethylationEPIC v1.0 BeadChip のゲノムワイドなバックボーンを基に構築されており、高い後方互換性を維持しながら(図2)、専門家のフォードバックとヒト癌および細胞サンプルのエピジェネティック評価に基づいて得られた新しいコンテンツを追加しています(表2、表3)。新バーションの
BeadChip では、DNA メチル化研究において、基礎となる一塩基多型(SNP)、クロスハイブリダイゼーション、およびマルチマッピングの挙動[8]により、日常的に除外される非機能性プローブを削除し、エピジェネティックス
コミュニティによって特定されたより多くの機能的コンテンツを搭載しました。
186,000 以上の新しいプローブが、ATAC-Seq (Assay for Transposable-Accessible Chromatin
using sequencing) および ChIP-Seq (chromatin immunoprecipitation sequencing)
実験によって同定された原発性腫瘍に関連する既知のエンハンサー、スーパーエンハンサー、CTCF 結合ドメイン、クロマチンのオープン領域をターゲットとしてデザインされました。
この新しいコンテンツは、主要なエピジェネティック研究者および最近の科学出版物によってアドバイスされました[9-14]。 |

図2. 従来の BeadChip との高い後方互換性
InfiniumTM MethylationEPIC v2.0 BeadChip は、InfiniumTM MethylationEPIC v1.0 および HumanMethylation450 BeadChip の既存の CpG バックボーンを基盤としています。 |
もう一つの改良点として、InfiniumTM MethylationEPIC v1.0 BeadChip ではカバーが不十分であった CpG アイランドとエクソンが、追加プロープによって徹底的にカバーされています。
さらに、新バージョンでは 450 以上の癌ドライバー変異が照会され、InfiniumTM MethylationEPIC v2.0 BeadChip は癌研究のためのマルチオミクスツールとなりました[15]。
あらゆる研究者は、将来のエピジェネティックな発見のための新しい最先端のコンテンツを活用できます。
InfiniumTM MethylationEPIC v2.0 BeadChip および v2.0 BeadChip のレガシーコンテンツ:
| ・ |
CpG アイランド |
| ・ |
ヒト幹細胞で同定された非 CpG サイトのメチル化領域(CHHサイト) |
| ・ |
ENCODE オープンクロマチンとエンハンサー |
| ・ |
FAMTOM5 エンハンサー |
| ・ |
DNase 高感受性部位 |
| ・ |
miRNA プロモーター領域 |
| ・ |
イルミナの Human Methylation450 BeadChip に搭載されているコンテンツの約85% |
InfiniumTM MethylationEPIC v2.0 BeadChip の新コンテンツ:
| ・ |
複数の癌について、腫瘍サンプルと正常サンプルで同定された異なるメチル化部位 |
| ・ |
ChIP-seq により同定された癌および細胞株サンプル中のエンハンサーおよびスーパーエンハンサー |
| ・ |
ATAC-seq によって特定された原発性ヒトがんにおいて識別的にアクセス可能なクロマチン領域 |
| ・ |
CpG アイランドのカバレッジ拡大 |
| ・ |
より正確なコピー数多型(CNV)検出のための強化されたエクソンカバレッジ |
| ・ |
一般的な癌ドライバー変異 |
|

 |
【2 種類の InfiniumTM ケミストリー】
|
InfiniumTM MethylationEPIC BeadChip は、業界で最も信頼性が高く、実績ある DNA 解析プラットフォームであるイルミナの InfiniumTM アッセイを使用しています。InfiniumTM メチル化アッセイは、DNA サンプル内の個々の CpG サイトを調べるためにデザインされた、ターゲット特異的な長鎖プローブが結合したビーズを使用しています。DNAメチル化は、バイサルファイト処理したゲノム
DNA の定量的ジェノタイピングにより測定されます。Infinium I アッセイおよび Infinium II アッセイが、お互いに補完的に働くことにより、広いターゲット領域をカバーします。
Infinium I アッセイ
|
Infinium I アッセイでは、1つの CpG サイトに対して、1個の「非メチル化」 DNA 検出プローブ及び 1 個の「メチル化」 DNA
検出用プローブの合わせて 2 個のプローブが利用されます。各プローブの 3' 末端は、保護されたシトシン(メチル化デザイン)、またはバイサルファイトによる変換と全ゲノムの増幅にって生成するチミン塩基(非メチル化デザイン)のいずれかにマッチする様にデザインされています。 |
Infinium II アッセイ
|
Infinium II アッセイは、1 個のターゲットサイトに対して、1 個のプローブのみを使用する様にデザインされています。プローブの 3'
末端は、検出サイトのすぐ上流の塩基に相補的で、1 塩基伸長の結果、「メチル化」 C または「非メチル化」 T に相補的な、標識された G または
A の塩基がそれぞれ付加されます。
Infinium II アッセイは、縮重オリゴヌクレオチドプローブを用いることにより、50mer の配列内に別の CpG サイトが存在したとしても、最大
3 箇所まで存在してもデータに影響を及ぼさず、ターゲットに隣接する CpG サイトのメチル化状態とは関係なく、検出サイトでのメチル化状態を評価することが可能です。 |
|
 |
【高精度で正確なメチル化データ】
|
Infinium アレイケミストリーでは、照会された各 CpG 部位に対して多数のビーズの複製が使用され、それぞれのビーズに数千のプローブが結合されています。その結果、Infinium
メチル化アッセイは非常に高精度なメチル化測定を提供します。
このことは、癌細胞株を用いた内部実験によって証明されており、技術的複製間で 99% 以上の再現性を示しました(図 3A)。
さらに、Infinium メチル化アッセイでは、1% 未満の偽陽性率で 0.2 のベータ値の差を検出することができるため、高い分析感度が達成されています。実験では、Infinium
MethylationEPIC v2.0 および v1.0 BeadChip のオーバーラップアッセイ(図 3B)とメチル化シークエンスデータ(図
3C)の高い相関性も示されました。Infinium BeadChips で得られる精度と正確さのレベルは、100x 以上の高いシークエンス深度でのみ得られることが研究で示されています[16]。
|
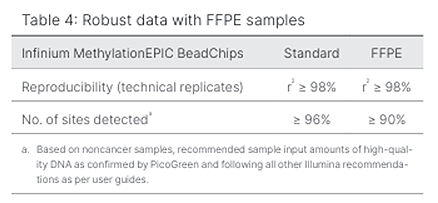
【FFPE サンプルへの適合性】
|
ホルマリン固定パラフィン包埋(FFPE)組織サンプルは、Infinium メチル化 HD アッセイの改良バージョンを使用すると堅牢なパフォーマンスを示します(表4)。
貴重なサンプルと最適なサンプルの完全性を実現するには、オプションの DNA Restoration 工程をお勧めします。
|
 |
【References】
|
| 1. |
Bibikova M, Lin Z, Zhou L, et al. High-throughput DNA
methylation profiling using universal bead arrays. Genome Res. 2006;16(3):383-393.
|
| 2. |
Fan JB, Oliphant A, Shen R, et al. Highly parallel SNP genotyping. Cold Spring
Harbor Symposia on Quantitative Biology. 2003;68:69-78.
|
| 3. |
Capper D, Jones DTW, Sill M, et al. DNA methylation-based
classification of central nervous system tumours. Nature. 2018;555(7697):469-474.
doi: 10.1038/nature26000.
|
| 4. |
Sengos AP, Aldape K. DNA Methylation Profiling: An Emerging Paradigm for
Cancer Diagnosis. Annu Rev Pathol. 2022;17:295321. doi:
10.1146/annurev-pathol-042220-022304.
|
| 5. |
Sadikovic B, Levy MA, Kerkhof J, et al. Clinical epigenomics:
genome-wide DNA methylation analysisfor the diagnosis of Mendelian disorders. Genet Med.
2021;23(6):1065-1074. doi: 10.1038/s41436-020-01096-4.
|
| 6. |
Horvath S, Raj K. DNA methylation-based biomarkers and the epigenetic
clock theory of ageing. Nat Rev Genet. 2018;19(6):371-384.
doi: 10.1038/s41576-018-0004-3.
|
| 7. |
Nwanaji-Enwerem JC, Colicino E. DNA Methylation–Based
Biomarkers of Environmental Exposuresfor Human Population Studies. Curr Environ
Health Rep. 2020;7(2):121-128. doi: 10.1007/s40572-020-00269-2.
|
| 8. |
Zhou W, Laird PW, Shen H. Comprehensive characterization, annotation and
innovative use of Infinium DNA methylation BeadChip probes. Nucleic Acids Res.
2017; 45(4):e22. doi: 10.1093/nar/gkw967
|
| 9. |
Corces MR, Granja JM, Shams S, et al. The chromatin accessibility
landscape of primary human cancers. Science. 2018;362(6413):eaav1898.
doi:10.1126/science.aav1898.
|
| 10. |
Chen H, Li C, Peng X, et al. A Pan-Cancer Analysis of Enhancer Expression in Nearly
9000 Patient Samples. Cell. 2018;173(2):386-399.
doi: 10.1016/j.cell.2018.03.027.
|
| 11. |
Lovén J, Hoke HA, Lin CY, et al. Selective inhibition of tumor
oncogenes by disruption of super-enhancers. Cell. 2013;153(2):320-334.
doi:10.1016/j.cell.2013.03.036.
|
| 12. |
Hnisz D, Abraham BJ, Lee TI, et al. Super-enhancers in the control
of cell identity and disease. Cell. 2013;155(4):934-47.
doi: 10.1016/j.cell.2013.09.053.
|
| 13. |
Jiang Y, Qian F, Bai X, et al. SEdb: a comprehensive human super-enhancer database.2019;47(D1):D235-D243.
doi: 10.1093/nar/gky1025.
|
| 14. |
Chapuy B, McKeown MR, Lin CY, et al. Discovery and characterization
of super-enhancer-associated dependencies in diffuse large B cell lymphoma. Cancer Cell.
2013;24(6):777-790. doi:10.1016/j.ccr.2013.11.003.
|
| 15. |
Bailey MH, Tokheim C, Porta-pardo E, et al. Comprehensive Characterization
of Cancer Driver Genes and Mutations. Cell. 2018;173(2):371-385.
doi: 10.1016/j.cell.2018.02.060.
|
| 16. |
Zhou L, Ng HK, Drautz-Moses DI, et al. Systematic evaluation of
library preparation methods and sequencing platforms for high-throughput whole
genome bisulfite sequencing. Sci Rep. 2019;;9(1):10383.
doi: 10.1038/s41598-019-46875-5.
|
|
|
 解析の流れ 解析の流れ
|
1. お見積もり
|
ご希望されるサンプル数などの情報を基に、価格・必要サンプル量などをご連絡いたします。
※正式なお見積もりは、代理店よりご提出いたします。 |
↓
2. サンプル送付
|
弊社宛てに、解析サンプルとQCシートを合わせてご送付ください。
送付された内容物を弊社で確認し、弊社から JS Link 社にサンプルを送付します。
| サンプル |
ご送付量 |
容量 |
濃度
*picogreen, Qubitなど |
O.D.260/280 |
O.D.260/230 |
| ゲノムDNA |
>1.5μg |
>30μL |
50ng/μL |
>1.7 |
>1.5 |
【ご送付サンプルについて】
|
解析サンプルをQCシートと同封の上、弊社までお送りください。
送付された内容物を弊社で確認し、弊社から JS Link 社にサンプルを送付します。
*本サービスは、海外での解析という性質上、サンプル受領後のキャンセルはお引き受けできません。
ご発送は、代理店へご依頼いただくか、直送願います。
発送の際の送料は、お客様負担となります。(着払いは受け取りできませんので、ご注意ください。)
*サンプルのご準備および送付方法に関する注意事項
|
| ・ |
サンプルの発送は、必ず、「サンプル送付方法およびご注意点(PDF)」をご参照の上、その内容に従って行ってください。 |
| ・ |
DNase、RNaseフリーのスクリューキャップ式の1.5mLエッペンチューブをご使用ください。 |
| ・ |
DNAサンプルは、電気泳動にて明瞭なバンドが検出され、分解がないことをご確認ください。サンプル送付時に、電気泳動写真を添付してください。 |
| ・ |
サンプル到着後、OD測定及び電気泳動による濃度・純度などの確認を行います。その結果によっては、DNAサンプルの再送をお願いする場合があります。予めご了承ください。
再度サンプルをお送り頂く場合、別途送料をご請求させて頂きます。 |
| ・ |
ゲノムDNAは、凍結融解をできるだけ避け、4℃でTE Bufferにて保存してください。長期保存の場合は、-20℃、又は、-80℃で保存してください。 |
| ・ |
送付の際は、チューブをパラフィルムで覆い、キャップの緩みや中身の漏れが起きない様にしてください。 |
| ・ |
サンプルの入ったチューブに油性マジックでサンプル名を明記し、遠心チューブやチューブボックス等に入れ、砕いたドライアイスとともに、梱包してください。DNAの場合は、アイスバックなどとともに梱包し、クール便でお送り下さい。ただし、DNAが冷凍状態で保存されている場合は、砕いたドライアイスとともに梱包して、クール便でお送り下さい。 |
| ・ |
組織・細胞をご送付の際には、ペレット状態にして、十分量のドライアイスを同梱の上、冷凍宅急便にて凍結状態にてご送付ください。 |
| ・ |
お預かりできるサンプルは、BSL2(バイオセーフティレベル2)までのものに限らせていただきます。感染性が著しく高いサンプル(HIV, HCV,
HBVウイルスに感染していることが確認されている患者由来の検体など)は、お受けできませんので、ご了承ください。 |
| ・ |
ヒト臨床サンプルの場合、インフォームドコンセントを得てからご提供願います。 |
| ・ |
ご提供頂くサンプル及び本作業から生じる知的財産権・工業所有権・安全性・インフォームドコンセントなどの問題については、弊社では、一切の責を負いかねます。 |
| ・ |
お送り頂いたサンプルは、ご返却いたしかねます。実験後のサンプルにつきましては、基本的には、弊社側にて破棄させて頂きますので、ご了承ください。 |
| ・ |
本事項を満たさない事で別途費用が発生した場合、お客様に費用のご負担をお願いすることがあります。 |
|
|
(サンプル送付先)
|
フィルジェン株式会社 受託解析部 宛
〒459-8011 名古屋市緑区定納山1丁目1409番地
Tel 052-624-4388 Fax 052-624-4389 |
|
|
|
3. サンプルの品質チェック
|
アガロースゲル、分光光度計などにより、品質(QC:Quality Control)チェック及び濃度測定を行います。
QCチェック結果が良好な場合は、引き続き解析を進めてまいります。
サンプル受領後、QCチェック結果が芳しくない場合は、以下のいずれかを選択してください。
a. 追加の精製/濃縮を行う(追加精製/濃縮で問題が解決される場合)。
この場合、サンプルの精製/濃縮料金が別途必要となります。
b. 再度新しいサンプルをご用意ください。
この場合、別途送料(海外)が必要となります。
c. お客様の同意の上、同じサンプルで実験を進めます。ただし、この場合、データの保証はされません。
結果如何によらず、通常料金をご請求させて頂きます。
*本サービスは、海外での解析という性質上、キャンセルはお引き受け出来ません。
やむを得ない理由で、キャンセルする場合は、それまでの工程に応じた料金をご請求させて頂きます。
↓ |
4. マイクロアレイ解析
|
以下は、Infiniumアッセイ及び、ハイブリダイゼーション、スキャンニング作業の手順です。
a. バイサルファイト処理
b. 全ゲノム増幅
c. 断片化処理
d. ビーズアレイとのハイブリダイゼーション
e. ポリメラーゼによる伸張反応
f. 蛍光標識
g. スキャンニング
↓ |
5. 解析データのご報告
|
データ解析結果を、CD-ROM/DVD/USBメモリーなどに収めて、ご報告します。
(納品物)
|
・Summary report(.pdf)
・Raw data(idat files, sample_sheet.csv)
・Unfiltered minfi Object(RGset.CS.rds): Objects available in the R environment
・Filtered minfi Object(RGset.GMS.rds): Objects available in the R environment
・Filtered beta (final.B_value.txt.gz)
・EPIC v2.0 probe annotation (EPICv2.probe.annotation.txt.gz) |
|
|
 価格/納期 価格/納期
|
【価格】
|
| サービス名 |
数量 |
税別単価 |
Cat.# |
Infinium® MethylationEPIC BeadChip
受託解析サービス |
8サンプル単位 |
お問い合わせください |
F-DN-EPIC-サンプル数 |
Infinium® Mouse MethylationEPIC BeadChip
受託解析サービス |
24サンプル単位 |
お問い合わせください |
F-DN-MEPIC-サンプル数 |
| * |
Infinium® MethylationEPIC BeadChip 受託解析サービスは、8サンプル単位での受注となります。 |
| * |
Infinium® Mouse Methylation BeadChip 受託解析サービスは、24サンプル単位での受注となります。 |
| * |
FFPE 由来サンプルの場合、Restore の費用が別途発生いたします。 |
|
【納期】
|
サンプル受領およびアレイ・試薬入荷後、5-6週間程度
※JS Link 社に、アレイ在庫がない場合は、アレイの入荷に別途 3 週間~ 1 ヶ月程度かかる場合があります。 |
|
本ページは、illumina社, JS Link 社のホームページに掲載されている情報を引用しています。
|
|