製品・サービス情報
|
|
BioTuring社製品 シングルセル研究パブリックデータ閲覧ソフトウェア
Talk2Data |
|
|
 |
 Talk2Data Talk2Data
| |
【公開されたシングルセル研究データの閲覧・再解析】
| |
Talk2Data では、公開されたシングルセル RNA-Seq データセットや空間オミックスデータセットを簡単に閲覧・再解析することができます。
細胞種などの情報は、論文情報などをもとにキュレーションされているため、論文を再現するためにアノテーションを作成する必要がありません。また、ダウンロードしたデータセットに対し、お客様側で新たにアノテーションを付与することも可能です。
さらに、閲覧データは、BBrowserX Pro や SpatialX と同じ様に下流分析まで実行することが可能です。これらの機能については、BBrowserX Pro、SpatialX の項目をご参照ください。計算・データ保存は、メーカークラウド上で行われます。 |
|
|
 |
| |
| |

メーカーの収集したパブリックデータの一覧
|
|

一覧から研究を選択すると、データセットへアクセスします。すでに著者の作成したアノテーションが紐づいています。 |
|
| |
【クロススタディ解析】
| |
BioTurning データベース全体における、複数の関心のある研究データをサブセット化して、カスタムアトラス(プロジェクト)を作成できます。データベースまたはアトラス作成後は、希少細胞集団の構成、DEG、パスウェイエンリッチメント解析、相関関係、マーカー遺伝子の検出といった下流解析を実行できます。
例)
| ・ |
パーキンソン病データセットにおいて、あらゆる細胞種および脳領域組織における SNCA 発現の特徴を明らかにする。 |
| ・ |
パーキンソン病とレビー小体型認知症(DLB)の脳のニューロンにおける DEG を検出する。 |
| ・ |
原発腫瘍部位の大腸がん T 細胞と隣接する正常組織の大腸がん T 細胞で、PDCD1 と共発現している遺伝子を見つける。 |
| ・ |
大腸がんの T 細胞と正常な大腸の T 細胞を区別するマーカー遺伝子を見つける。 |
|
|
| |
| |
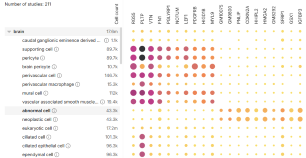
Composition グラフ
データセット内の遺伝子発現を細胞種、条件、組織など、様々な観点から探索することができます。階層的な可視化によってデータはツリー構造で整理され、段階的に絞り込むことが可能です。例えば、「組織」から「細胞種」へ、さらに「条件」へと枝分けれしていくことで、各階層における詳細な遺伝子発現パターンを確認できます。 |
|

Differential expression analysis
2 つの細胞集団の発現プロファイルを比較し、発現レベルが異なる遺伝子(DEG)を特定します。高度なフィルタリングを活用することで、注目したい細胞群同士を比較できます。 |
|
 デモ版ダウンロード デモ版ダウンロード
|
弊社( )までお問合せください。 )までお問合せください。 |
 システム要件/サポートされる生物種 システム要件/サポートされる生物種
|
ウェブブラウザ上で動作します。
計算・データ保存は、メーカーのクラウド上で行われます。
インターネット接続を必要とします(Google Chrome 推奨)
接続について、デモ版にてテストすることを推奨しております。
生物種は、ヒトおよびマウスがサポートされています。 |
 技術資料・セミナー 技術資料・セミナー
|
フィルジェンWEBセミナー
| 公開シングルセル RNA-Seq データの再解析「皮膚がんのリスクとしての結節性痒疹の探索」(2024年11月14日) |
 |
| 公開シングルセルデータ・空間オミックスデータセットのインタラクティブな探索(2023年11月29日) |
 |
|
 価格 価格
|
アカデミックライセンス
| 商品名 |
ライセンスタイプ |
税別価格 |
カタログ# |
| Talk2Data |
アカデミック、1年間 1ユーザー 指定ライセンス |
お問合せ |
F-BTD |
コマーシャル
| 商品名 |
ライセンスタイプ |
税別価格 |
カタログ# |
| BioTuring ecosystem |
コマーシャル、年間ライセンス |
お問合せ |
お問合せ |
| * |
本ソフトウェアは、年間ライセンスです。 |
| * |
解析にはインターネット接続が必要です。 |
| * |
テクニカルサポートは、電話、あるいは、電子メールでの対応のみです。 |
| * |
データセットのエクスポートやアトラス作成に一部制限があります。 |
|
例)アカデミックライセンスの場合: 1年間で最大100,000のエクスポート、セルアトラス機能で最大30,000細胞/1解析のカスタムアトラスを作成 |
| * |
BioTuring社製品を複数モジュール組み合わせてご購入の場合ディスカウントがございます。 |
| * |
共通機器利用向けのライセンスもございます。詳細はお問合せください。 |
| * |
コマーシャルライセンスの仕様については、お問合せください。 |
|
|